作者简介:王宪龙(1976—),男,本科,副主任技师,研究方向:微生物检验。
目的 了解济宁市一起聚集流感疫情的甲型H1N1流感病毒全基因组特征,为甲型H1N1流感防控提供依据。方法 对在一起聚集流感疫情采集的12份患者咽拭子标本进行流感病毒实时荧光定量PCR法核酸检测,对阳性标本进行病毒培养和全基因组测序,利用DNASTAR软件分析核苷酸和氨基酸同源性,利用MEGA软件构建进化树,利用NetNGlyc、GPS-SUMO、NetPhos软件分别分析糖基化位点、类泛素蛋白修饰分子化位点和磷酸化位点。结果 12份咽拭子标本中,4份标本甲型H1N1流感病毒核酸检测阳性,分离流感病毒株4株。同2018—2019年度北半球疫苗株A/Brisbane/02/2018相比,8个基因节段核苷酸同源性范围为98.5%~99.8%;编码的10种蛋白氨基酸同源性范围为98.2%~100.0%。在进化树上,4株毒株位于两个进化簇,其中3株毒株位于同一进化簇,1株毒株位于不同进化簇。4株毒株编码的10种蛋白共发生50处氨基酸位点置换,血凝素蛋白抗原表位2个氨基酸位点发生置换,神经氨酸酶蛋白酶活性位点和神经氨酸酶抑制剂耐药位点均未发生变异,聚合酶酸性蛋白、聚合酶碱性蛋白1和聚合酶碱性蛋白2抑制剂耐药位点均未发生变异。结论 本次由甲型H1N1流感病毒引起学校流感聚集疫情有2个不同的源头,应加强对学校流感聚集疫情监测,加强学校流感防控。
Objective To analyze the whole genome characteristic of influenza A(H1N1) pdm09 virus outbreak in a school in Jining, so as to serve as a reference for prevention and control of influenza A (H1N1) pdm09.M-ethods Real time fluorescence quantitative PCR were used to detected the 12 nasopharyngeal swab specimens from patients. The positive specimens were cultured and sequenced the whole genome. DNASTAR was used to analyze the homology. MEGA was used to construct the phylogenetic tree. NetNGly, GPS-SUMO, NetPhos were used to predict the glycosylation sites, the small ubiquitin-like modifier sites and the phosphorylation sites respectively.Results Among of 12 specimens, 4 specimens were showed positive for influenza A (H1N1) pdm09 virus nucleic acid, and 4 virus strains were isolated. Compared to the vaccine strain A/Brisbane/02/2018, the homology of nucleic acids of 8 gene segments was 98.5%-99.8%. The homology of amino acids of 10 proteins was 98.2%-100%. In the evolutionary tree analysis, four strains were located in two evolutionary clusters, three strains in the same evolutionary cluster and one strain in a different evolutionary cluster. A total of 50 amino acid site substitutions occurred in the 10 proteins encoded by the four strains, including two amino acid sites in the hemagglutinin antigen epitope, and there was no mutation in the neuraminidase (NA) protease active site, polymerase acidic protein (PA), polymerase basic protein 1 (PB1) and polymerase basic protein 2 (PB2) inhibitor resistance sites, and neuraminidase inhibitor resistance sites. In addition, the glycosylation sites and SUMO sites were not mutated, while multiple phosphorylation sites were substituted.Conclusions There are 2 different sources of influenza A (H1N1) virus causing the school influenza outbreak. School influenza outbreak surveillance and school influenza prevention and control should be strengthened should be enhanced.
流行性感冒病毒(Influenza virus)属正粘病毒科, 主要通过呼吸道传播, 我国北方秋冬季高发。流感病毒主要分甲型、乙型和丙型, 人主要感染甲型和乙型, 包括甲型H1N1、季节性H3N2、BVictoria 系和B Yamagata 系流感病毒。甲型H1N1流感自2009年在墨西哥暴发随后引起世界大流行, 甲型H1N1流感病毒全基因组包含8个基因节段, 由人流感、猪流感和禽流感病毒重组而来, 可编码10种以上的蛋白质[1]。目前甲型H1N1流感是我国和济宁市主要的季节性流感之一[2, 3, 4, 5]。2019年1月山东省某县某学校发生一起流感样病例聚集疫情, 为了解病原特征, 本研究开展了核酸检测、流感病毒分离和全基因组测序, 为学校制定甲型H1N1流感防控方案提供基础数据支持。
1.1.1 样品来源 采集2019年1月7日山东省济宁市某学校聚集疫情中流感样病例(体温≥ 38 ℃, 并伴有咳嗽或咽痛之一者)的咽拭子标本12份。
1.1.2 仪器和试剂 MagNA Pure LC2.0全自动核酸提取仪和LightCycler 480 II实时荧光定量PCR仪均购自Roche公司; CO2培养箱购自Thermo公司; 生物安全柜购自上海力申科学仪器有限公司; 病毒采样管(MT0301)购自友康生物科技(北京)股份有限公司; MagNA Pure LC Total Nucleic Acid Isolation Kit核酸提取试剂盒(货号:03038505001)购自Roche公司; 甲型H1N1流感病毒(2009)RNA检测试剂盒(PCR-荧光探针法)(货号:DA2391)购自达安基因股份有限公司; 流感病毒鉴定试剂盒由国家流感中心提供。
1.2.1 核酸检测和病毒分离 利用全自动核酸提取仪提取200 μ L咽拭子标本核酸, 取5 μ L核酸进行甲型H1N1流感病毒实时荧光定量PCR检测, 核酸提取和核酸检测详见商品化试剂盒说明书。37 ℃, 5%CO2培养MDCK狗肾细胞, 待细胞汇合度达90%后接种阳性咽拭子标本1 mL, 培养3~4 d后进行HA血凝实验。流感病毒细胞培养详见本实验室之前的描述[6--5]。
1.2.2 基因测序和生物信息学分析 对分离的毒株送上海伯杰医疗科技有限公司进行甲型H1N1流感病毒全基因组测序。采用DNASTAR7.0.1软件进行同源性分析、MEGA 7.0.14软件构建进化树、NetNGlyc-1.0分析糖基化位点、GPS-SUMO分析类泛素蛋白修饰分子(small ubiquitin-like modifier, SUMO)化位点和NetPhos-3.1分析磷酸化位点。
2019年1月7— 10日, 在济宁市某学校发生一起流感样病例聚集疫情, 该校累计报道流感病例74例, 其中1月7日22例, 1月8日18例, 1月9日25例, 1月10日9例。全学校流感样病例发病率为2.05%(74/3 610), 分布在10个班级, 年龄12~14岁, 男56例, 女18例。临床表现:体温37.5 ℃~40.0 ℃, 伴有咳嗽、咽痛, 无住院、重症病例。均无流感疫苗接种史和病死禽畜接触史。
依据《全国流感监测方案(2017年版)》每起暴发疫情采集约10份标本的要求, 采集该校流感样病例咽拭子标本 12份, 进行流感病毒核酸检测, 结果4份标本甲型H1N1流感病毒阳性, 阳性率为33.33%。对4份甲型H1N1流感病毒核酸检测阳性标本进行病毒培养, 培养出流感病毒4株, 分别命名为A/shandongweishan/SWL32/2019(H1)、A/shandongweishan/SWL33/2019(H1)、A/s handongweishan/SWL35/2019(H1)、A/shandongweisha n/SWL312/2019(H1)。
对分离4株毒株进行全基因组测序, 4株毒株均获得甲型H1N1流感病毒血凝素蛋白(hemagglutinin, HA)、神经氨酸酶(neuraminidase, NA)、基质蛋白(matrix protein, MP)、非结构蛋白(non-structural protein, NS)、核蛋白(nucleoprotein, NP)、聚合酶酸性蛋白(polymerase acidic protein, PA)、聚合酶碱性蛋白1 (polymerase basic protein 1, PB1)和聚合酶碱性蛋白2(polymerase basic protein 2, PB2)等8个节段全基因组序列, 获得基因核苷酸长度分别为1 701 bp、1 410 bp、982 bp、838 bp、 1 497 bp、2 151 bp、2 274 bp和2 280 bp。同2018— 2019年度北半球疫苗株A/Brisbane/02/2018(GISAID编号:EPI_ISL_330190)相比, HA、NA、MP、NS、NP、PA、PB1和PB2基因核苷酸同源性范围分别是98.5%~98.9%、99.0%~99.3%、99.2%、99.4%~99.8%、99.1%~99.4%、99.2%~99.4%、99.3%~99.4%、99.4%~99.5%; HA、NA、基质蛋白1(matrix protein 1, M1)、基质蛋白2(matrix protein 2, M2)、非结构蛋白1(non-structural protein 1, NS1)、核输出蛋白(nuclear export protein, NEP)、NP、PA、PB1和PB2蛋白氨基酸同源性范围分别98.2%~98.4%、98.7%~99.1%、100.0%、97.9%、99.1%~99.5%、99.2%~100%、99.8%、99.4%~99.9%、99.5%~99.6%和99.5%~99.7%。
利用MEGA 7.0.14软件, 采用邻位归并法(neighbour-joining)的Kimura 2-parameter model 模型构建HA、NA、MP、NS、NP、PA、PB1和PB2基因进化树。4株毒株的8个基因均与疫苗株A/Brisbane/02/2018均位于同一分支。然而, 毒株A/shandongweishan/SWL33/2019(H1)、A/shandongweishan/SWL35/2019(H1)、A/shandongweishan/SWL312/2019(H1)的8个基因均位于同一进化簇, 毒株A/shandongweishan/SWL32/2019(H1)的8个基因均位于另一进化簇。此外, 在HA进化树中4株济宁分离株与疫苗株A/Brisbane/02/2018共同属于6B.1分支, 见图1。
与疫苗株A/Brisbane/02/2018相比, HA蛋白发生15处氨基酸变异, 4株毒株HA蛋白受体结合位点130 环( 135~138)、190螺旋(190-198)和220 环(221~228)均未发生变异; 毒株A/shandongweishan/SWL33/2019(H1)、A/shandongweishan/SWL35/2019(H1)、A/shandongweishan/SWL312/2019(H1)在抗原决定簇Sb(184-195)发生了T185I置换, 毒株A/shandongweishan/SWL32/2019(H1)在抗原决定簇Ca2(137-142、221-222)发生了A141T置换。NA、M2、NS1、NEP、NP、PA、PB1、PB2蛋白分别发生9、3、3、3、2、4、6、5处氨基酸置换。见表1。此外, 4株毒株均未发生E119A/D/G/V、Q136K/R、I223K/R/V/T、S247N/G/R等导致NA抑制剂耐药的变异; 均未发生K229R等导致PB1抑制剂耐药的变异; 均未发生Q306H、S324I/N/R、F404Y和 N510T等导致PB2抑制剂耐药的变异。4株毒株和疫苗株的M2蛋白31位氨基酸均为N, 说明均对金刚烷胺耐药。
| 表1 氨基酸变异位点分析 Table 1 Analysis of amino acid variation sites |
利用NetNGlyc-1.0分析糖基化位点, 济宁4株毒株蛋白的糖基化位点均未发生变异。利用GPS-SUMO分析SUMO化位点, 济宁4株毒株的M1、NS1、NEP、NP、PB1、PB2均未发生SUMO化位点变异。利用NetPhos-3.1分析磷酸化位点, A/shandongweishan/SWL32/2019(H1)的HA蛋白增加T141、T302、S496磷酸化位点、丢失110位磷酸化位点, NEP蛋白增加T89磷酸化位点, NP蛋白增加S498磷酸化位点。毒株A/shandongweishan/SWL33/2019(H1)、A/shandongweishan/SWL35/2019(H1)、A/shandongweishan/SWL312/2019(H1)的HA蛋白丢失185位磷酸化位点, NS1蛋白增加T155磷酸化位点。4株毒株的PA蛋白均丢失61位磷酸化位点。
结合本次聚集疫情的核酸检测、病毒分离和全基因组测序结果, 确定引起本次学校流感样病例聚集疫情的病原体是甲型H1N1流感病毒, 共确诊病例4例, 分离甲型H1N1流感病毒毒株4株, 与甲型H1N1流感是济宁市2019年主要流行的季节性流感一致[4]。同疫苗株A/Brisbane/02/2018相比, 4株毒株8个基因节段核苷酸同源性范围为98.5%~99.8%, 其中HA基因同源性最低, 与HA容易发生变异一致; 4株毒株编码10个蛋白氨基酸同源性范围为98.2%~100.0%, 其中HA蛋白同源性最低, M1蛋白同源性最高为100.0%。
全基因组进化分析显示, 在进化树上, 4株毒株8个基因片段与疫苗株A/Brisbane/02/2018均位于6B.1进化分支, 说明8个基因片段与疫苗株A/Brisbane/02/2018在进化上亲缘较近。与济宁市2018— 2019年甲型H1N1流感病毒均位于6B.1进化分支结果一致[4], 进一步说明位于6B.1进化分支甲型H1N1流感病毒是济宁市2018— 2019年的主要流行株。本研究4株毒株8个基因片段均未发生基因重组现象。然而, 毒株A/shandongweishan/SWL33/2019(H1)、A/shandongweishan/SWL35/2019(H1)和A/shandongweishan/SWL312/2019(H1)8个基因片段与毒株A/shandongweishan/SWL32/2019(H1)8个基因片段位于不同的进化簇, 说明导致本次学校甲型H1N1流感疫情可能至少有两个传染源和两条传播途径。本次疫情不同来源的流感病毒可能在相近的时间段, 通过不同的学生传入学校, 学生在上学、课间、放学期间人员接触密切, 流感容易扩散, 不同来源的流感病毒可能在不同的班级交互传染, 最终造成此次共10个班级共74人发病的流感疫情。
HA是甲型H1N1流感病毒的受体结合位点和最重要的表面抗原[7, 8]。本研究4株毒株HA受体结合位点均未发生变异, 说明4株毒株对宿主受体的结合影响不大。HA共含有4个抗原决定簇。在HA抗原决定簇发生4个以上氨基酸变异且至少分布在2个以上的抗原决定簇即具有流行病学意义的新变种[9, 10], 本研究4株毒株HA抗原决定簇均仅发生了1个氨基酸位点的变异, 说明造成本次学校疫情的甲型H1N1流感病毒不是具有流行病学意义的新变种。
NA是目前流感最重要的治疗靶点, NA抑制剂是目前临床治疗流感最重要的药物[11, 12]。然而随着流感的不断进化变异, 湖北省、包头市等地区2019年甲型H1N1流感病毒发现了NA抑制剂耐药变异[13, 14]。本研究4株毒株NA蛋白共发生9处氨基酸置换, 但NA活性位点和NA抑制剂耐药位点均未发生变异, 与2019年济宁市甲型H1N1流感病毒哨点医院监测结果类似[4], 说明对奥司他韦等NA抑制剂药物敏感。M2是甲型流感的治疗靶点, 金刚烷胺等烷胺类药物通过干扰M2抑制流感复制, 流感病毒极易产生金刚烷胺的耐药变异[15], 本研究4株毒株均为金刚烷胺耐药株, 进一步说明目前金刚烷胺不适合用于流感的治疗。
PA、PB1和PB2构成流感病毒的聚合酶, PA抑制剂巴洛沙韦、PB1抑制剂法匹拉韦、PB2抑制剂吡莫地韦等是近年出现的新型抗流感特异性药物[16]。虽4株毒株的PA、PB1和PB2发生多处氨基酸置换, 但未发现PA、PB1和PB2抑制剂耐药位点的变异, 说明4株毒株对巴洛沙韦、法匹拉韦、吡莫地韦等聚合酶抑制剂药物敏感。
蛋白翻译后修饰在流感病毒蛋白维持正常分子结构和生物学功能发挥关键作用, 流感病毒编码蛋白糖基化改变能够影响其分子构象、受体结合能力、免疫原性、神经氨酸酶活性、复制能力和致病能力等[17]。本研究4株毒株编码的10种蛋白均未发生糖基化位点的变异。研究表明流感病毒M1、NS1、NEP、NP、PB1和PB2能够SUMO化修饰, 这些蛋白的SUMO化修饰广泛参与流感病毒的抗宿主免疫、病毒复制、病毒组装、病毒增殖等过程[18]。本研究4株毒株SUMO化位点未发生变异。流感病毒编码的10种蛋白均能够被磷酸化, 磷酸化在流感病毒感染、蛋白核定位、蛋白聚合等多种生物学过程[19]。本研究4株毒株编码的HA、NS1、NEP、NP、PA磷酸化位点发生置换, 这些磷酸化位点的改变可能影响相关蛋白的结构和生物学功能, 但需要进一步研究确定。
学校人员密集, 学生密切接触频繁, 学校是流感聚集疫情的高发地点。本研究提示学校流感聚集疫情复杂多样, 可能发生有两个以上不同源头引起的聚集疫情, 因此应该加强对学校流感聚集疫情的监测力度, 为学校聚集疫情的有效防控提供参考。
利益冲突声明 所有作者声明不存在利益冲突
编辑:符式刚
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|



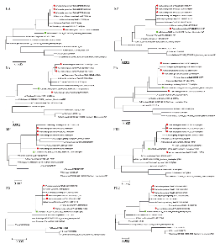
 表示疫苗株B/Washington/02/2019;
表示疫苗株B/Washington/02/2019;  表示本次聚集疫情流感病毒分离株。
表示本次聚集疫情流感病毒分离株。