作者简介:欧新华(1971—),女,本科,主任技师,研究方向:微生物病原学检测与基因组学研究。
目的 对长沙市2020年急性胃肠炎暴发疫情中诺如病毒GⅡ.2[P16]型进行全基因组序列测定以及遗传进化特征分析。方法 采集2020年诺如病毒暴发疫情标本,采用逆转录聚合酶链反应(RT-PCR)方法扩增开放阅读框(ORF)1和ORF 2连接区进行分型鉴定。GⅡ.2[P16]型阳性标本在二代测序Miseq平台进行测序。测序结果进行序列比对及进化分析。结果 2020年本市共发生诺如病毒暴发疫情29起,共采集标本277份,阳性检出率为44.04%(122/277)。分型鉴定发现诺如病毒GⅡ.2[P16]型占比45.08%(55/122)。从疫情分布月份和机构来看,9—12月为暴发高峰,疫情发生在幼儿园和学校占比89.66%(26/29)。对53份GⅡ.2[P16]型ORF1和ORF2连接区序列进行进化树分析发现毒株处于多个分支,序列之间的同源性为98.5%~100%。对4株GⅡ.2[P16]型全基因组序列进行同源性比较发现毒株之间的同源性为98.5%~99.5%,与GⅡ.2[P16]2016-2017(KY771081)毒株同源性为97.9%~98.2%,进化树处于不同亚分支。RdRp区域氨基酸位点发生T396A和T464A突变,HBGA结合位点保守未发生突变。结论 长沙地区诺如病毒暴发疫情中以GⅡ.2[P16]型突变株传播为主,毒株由GⅡ.2[P16]2016-2017持续传播进化而来,并进化为另一个分支。本地区暴发的诺如病毒疫情主要发生在学校以及幼托机构,应持续加强对诺如病毒的监测。
Objective To investigate the genetic evolutionary characteristics of norovirus GⅡ.2[P16] type from acute gastroenteritis outbreaks in Changsha city in 2020 by whole genome sequencing.Methods Norovirus outbreak samples were collected in 2020 year. The open reading frame (ORF) 1 and ORF 2 junction regions were amplified by reverse transcription polymerase chain reaction (RT-PCR) for further norovirus genotyping. The full-genome of GⅡ.2[P16] positive samples were obtained from Miseq platform (Illumina). Sequence alignment and evolutionary analysis were performed.Results There were 29 outbreaks of norovirus in Changsha city in 2020. 277 samples were collected, and the positive rate was 44.04% (122/277). Genotype identification showed that GⅡ.2[P16] norovirus was accounted for 45.08% (55/122). The peak of norovirus pandemic appeared from September to December. Kindergarten and school of the outbreak places were accounted for 89.66% (26/29). Based on ORF1 and ORF2 junction nucleotide sequence, phylogenetic tree analysis revealed that 53 norovirus strains distributed in different branches. Those strains shared 98.5%-100% similarity with each other. The 4 strains of full-genome GⅡ.2[P16] sequences showed 98.5%-99.5% similarity with each other, and shared 97.9%-98.2% similarity with GⅡ.2[P16]2016-2017(KY771081) strain. Phylogenetic tree showed the 4 strains in this research belong to different sub-branches. The amino acid sites in RdRp region contain T396A and T464A mutations. And the HBGA binding sites in GⅡ.2[P16] viruses were conserved.Conclusions The spread of GⅡ.2[P16] type mutant strain is predominant in the norovirus outbreak in Changsha city, which evolved from GⅡ.2[P16]2016-2017 and have evolved into a different branch. Outbreaks are mainly reported in kindergarten and school. Our findings have improved the general understanding of the evolution, genetic diversity, and wide distribution of noroviruses.
诺如病毒是引发急性胃肠炎暴发疫情的主要病原体, 属于杯状病毒科诺如病毒属。诺如病毒造成的感染疫情大多发生在冬春季节, 人群被感染后的主要症状为腹泻、发热、呕吐等, 多为急性感染。目前能导致感染性腹泻疫情的诺如病毒病原体分为10种基因群(GⅠ 至GⅩ ), 其中诺如病毒基因群Ⅰ 的GⅠ 型和基因群Ⅱ 的GⅡ 型是造成暴发疫情的主要病原体。自2002年以来, GⅡ .4型诺如病毒在全世界造成了大范围流行, 并每隔2~3年重组为新毒株造成持续流行[1]。2014年GⅡ .17[P17]型诺如病毒替代GⅡ .4型成为我国暴发疫情优势毒株[2]。2016年以来, 诺如病毒GⅡ .2[P16]重组毒株(以下简称GⅡ .2[P16]2016-2017)是造成北京、湖南、深圳等地暴发疫情的主要病原体[3, 4, 5, 6, 7, 8]。近年来, 长沙市感染性腹泻暴发疫情中GⅡ .2[P16]、GI.6[P11]、GⅡ .3[P12]、GⅡ .13[P21]等多种基因型别被检测到[9, 10, 11]。本文旨在分析2020年长沙市诺如病毒暴发疫情中病原谱的构成, 以及GⅡ .2[P16]型的全基因组序列特征及遗传进化分析。
感染性腹泻暴发疫情定义为同一集体单位3 d内发生3例以上急性胃肠炎病例。按照《感染性腹泻诊断标准》(WS271-2007)收集2020年长沙市感染性腹泻疫情中的肛拭子、粪便标本, 采集后于4 ℃下运输至本实验室。未及时处理的标本暂存于-80 ℃超低温冰箱。流行病学资料来源于中国疾病预防控制信息系统。
取100 mg粪便标本重悬于1 mL PBS溶液, 制成悬浮液。肛拭子与悬浮液标本进行10 000 r/min离心后, 取上清液进行磁珠法核酸提取(西安天隆)。采用诺如病毒GI/GⅡ 型核酸荧光检测试剂盒(江苏硕世)进行检测, 选取CT值小于30的标本进行后续的测序分析。
采用RT-PCR方法对诺如病毒阳性标本进行分型鉴定, 引物为G2SKF/G2SKR[12]。病毒序列通过诺如病毒在线基因分型工具Norovirus Genotyping Tool(http://www.rivm.nl/mpf/norovirus/typingtool)进行分型。选取CT值小于20的标本进行二代测序分析。毒株核酸RNA提取后, 采用诺如病毒GⅡ 型全基因组捕获试剂盒扩增基因组全长(北京微未来)。扩增产物纯化后先用Qubit 2.0进行核酸浓度定量, 二代测序建库核酸起始量为1 ng DNA, 建库采用Nextera XT DNA 文库构建试剂盒(Illumina, USA)。文库构建完成后用Miseq v2 试剂盒(Illumina, USA)在Miseq平台上进行测序。下机数据先用Fastqc和Cutadapt进行数据质量分析和去接头, 再用Virus Identification Pipeline软件进行比对分析和过滤, 处理后的数据用SPAdes-3.13.0软件进行序列拼接与组装。
序列先用Lasergene进行序列比对, 采用MEGA 6.0软件构建进化树, 方法为邻接法(N-J算法), bootstrap值为1 000。氨基酸位点分析采用BioEdit软件。
2020年长沙市共监测到29起诺如病毒引起的暴发疫情, 总计波及1 524人次, 共采集标本277份, 诺如病毒核酸检测阳性122份, 阳性率为44.04%(122/277)。分型鉴定发现诺如病毒GⅡ .2[P16]型占比45.08%(55/122), GⅡ .4[P31]型占比6.56%(8/122), GI.3[P13]型占比4.10%(5/122), GI.3[P10]型占比2.46%(3/122), GI.6[P11]占比0.82(1/122), GⅡ .6[P7]型占比0.82(1/122), 余下49份为诺如病毒核酸检测阳性, 但CT值太高, 未进一步鉴定分型。从疫情分布月份来看, 9— 12月为暴发高峰, 共发生25起; 其次为4— 6月发生4起, 为次高峰。幼儿园和学校机构发生26起, 其他机构发生3起。其中由GⅡ .2[P16]型造成的诺如病毒暴发疫情有24起发生在幼儿园和学校机构, 由GⅡ .6[P7]型和GⅡ .4[P31]型造成的幼儿园诺如病毒暴发疫情各1起。秋冬季为本市诺如病毒疫情高发季节。
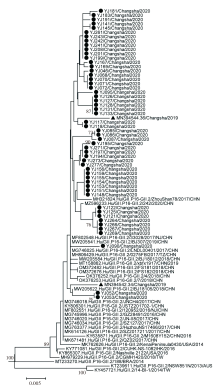
本研究共获得53份诺如病毒GⅡ .2[P16]型开放阅读框(ORF)1和ORF2连接区序列。进化树分析发现序列处于不同分支(图1), 序列之间的同源性为98.5%~100%。
本研究获得4株诺如病毒GⅡ .2[P16]型全基因组序列, 毒株分别位于4起暴发疫情中, 序列提交至GenBank数据库, 编号为OL826940~OL826943。对全基因组核苷酸序列进行同源性比较发现, 与上海地区发现的毒株(MT158862)同源性为99.0%~99.3%, 与北京地区发现的毒株(MW205584)同源性为99.1%~99.4%, 与香港2016年的GⅡ .2[P16](KY771081)毒株同源性为97.9%~98.2%, 与2019年长沙本地毒株(MN394544和MN394542)同源性为97.7%~98%。毒株之间的同源性为98.5%~99.5%。
RdRp核苷酸序列与北京地区2020年发现的GⅡ .4[P16]型(OL336338)同源性为98.4%~99.7%, VP1核苷酸序列与上海地区2019年发现的GⅡ .2[P16]毒株(MT158862)的同源性为99.0%~99.3%。
基于核苷酸序列进化树分析发现, RdRp区在进化分支上与北京地区(OL336338、OL336332)以及日本(LC646332、LC646333)发现的毒株位于同一分支。VP1序列进化树发现, 与日本(LC646332), 深圳地区(MK614128)和上海地区(MT158862)发现的毒株亲缘关系较近。与长沙市2019年的诺如病毒毒株处于不同分支(图2)。
对4条GⅡ .2[P16]型全基因组序列进行分析发现, VP1核苷酸序列存在G1218T突变, HBGA结合位点氨基酸序列保守未发生突变, 存在V256I突变。RdRp核苷酸序列发生A315G、A801C、T846C、C867T、C1386T和A1390G位点突变, 氨基酸序列中存在GⅡ .2[P16]2016-2017型毒株携带的突变位点, 为S293T、V332I、K357Q和T360A突变, 其中YJ070/Changsha/2020序列RdRp区域发 现T396A和T464A突变, 5份毒株序列具有T464A突变。
诺如病毒GⅠ 和GⅡ 型中绝大部分基因型均能感染人类, 相较于GⅠ 型, GⅡ 型是历年来造成急性胃肠炎疫情的主要病原体[1], 以儿童感染为主[13, 14]。基因重组是诺如病毒基因组常见的现象, 基因重组后产生的新型毒株在全世界引起了大范围的腹泻暴发疫情, 造成巨大的经济损失, 如GⅡ .17[P17]、GⅡ .4[P31]、GⅡ .2[P16]等[3, 9, 15]。本研究对2020年长沙市急性胃肠炎暴发疫情中的诺如病毒进行鉴定分析, 对GⅡ .2[P16]型诺如病毒进行全基因组序列分析, 有助于掌握长沙市诺如病毒的遗传进化特征。
从2020年的病原谱结果来看, 多种诺如病毒型别在长沙市造成了暴发流行, 但暴发疫情中GⅡ .2[P16]型仍然为优势毒株(占比45.08%)。从2016年开始GⅡ .2[P16]成为我国诺如病毒疫情主要基因型, 湖南省2017年开始暴发疫情中以GⅡ .2[P16]基因型为主, 本研究与相关报道相一致[4, 15, 16, 17, 18]。全基因组序列分析发现毒株分别与来自上海, 北京, 香港等地区的毒株亲缘关系接近, 进一步表明本市由于交通便利, 人口流动增强, 诺如病毒可以快速地在地域之间进行传播。RdRp核苷酸序列与北京地区2020年发现的毒株(OL336338)同源性为98.4%~99.7%, 表明诺如病毒通过基因重组突破宿主免疫屏障造成了不同地域诺如病毒的暴发流行[19]。基因组序列进化树分析发现ORF1和ORF2连接区序列分布于多个分支, 说明长沙市存在诺如病毒的多条传播链。
有研究表明RdRp序列位点的突变可能是造成GⅡ .2[P16]持续流行的重要角色[20]。Tohma等[20]对2016年冬季暴发流行的GⅡ .2[P16]2016-2017新变异毒株进行研究发现, 毒株RdRp区域携带了氨基酸S293T、V332I、K357Q和T360A突变, 这些突变位点与持续流行相关。本研究中的全基因组序列, RdRp区域包含上述氨基酸突变位点, 并发生了T396A和T464A位点突变, 该突变位点是否对GⅡ .2[P16]的传播能力造成影响还有待进一步研究。Ao等[15]对全国的GⅡ .2[P16]2016-2017型毒株进行研究指出2016年11月之后出现的V256I突变位点, 能使GⅡ .2[P16]2016-2017型毒株额外获得对A型分泌型唾液的结合能力, 进一步造成了毒株的传播。长沙地区诺如病毒GⅡ .2[P16]型的VP1氨基酸序列保守, HBGA结合位点氨基酸未发生突变。但氨基酸序列存在V256I突变, 使毒株能结合多种型别的唾液酸, 增强了毒株的传播能力。
另一方面RdRp和VP1序列与GⅡ .2[P16]2016-2017毒株在进化树处于不同亚分支, 但分析却显示与GⅡ .2[P16]2016-2017毒株同源性最高。说明长沙地区GⅡ .2[P16]2020毒株, 由GⅡ .2[P16]2016-2017毒株进化而来, 但已进化为另一分支。
综上所述, 长沙地区2020年诺如病毒暴发疫情虽存在多种型别流行, 但以GⅡ .2[P16]型传播为主, 由GⅡ .2[P16]2016-2017毒株持续进化流行而来, 存在多个氨基酸突变位点, 具备持续感染的能力, 应继续当加强长沙诺如病毒的监测, 及时发现新出现的诺如病毒新型别, 预防病毒性腹泻暴发。
利益冲突声明 所有作者声明不存在利益冲突
编辑:王佳燕
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|