作者简介:朱燕涛(1997—),男,硕士研究生,研究方向:分子流行病。刘俊仪(1999—),女,硕士研究生,研究方向:分子流行病。#刘俊仪同为第一作者
目的 基于丙型肝炎病毒(hepatitis C virus, HCV)核衣壳蛋白( nucleocapsid protein)基因序列了解昆明地区人类免疫缺陷病毒(human immunodeficiency virus, HIV)/HCV共感染患者中HCV基因型及基因亚型流行情况。方法 2019年3—8月在云南省传染病医院收集临床诊断为HIV/HCV共感染住院且户籍地为昆明所辖14个县级市、区及县的患者血清,从血清中提取病毒RNA,反转录合成第一链cDNA,采用HCV核衣壳蛋白基因特异性引物进行巢式PCR扩增。结果 共收集到临床诊断疑似HIV/HCV患者样本64例,采用HCV核衣壳蛋白基因特异性引物扩增,其中17例样品扩增阳性。序列分析结果显示,有9例病例的序列与HCV 3b基因亚型序列位于同一个进化分支,核苷酸同源性为 93.3%~95.2%;5例病例的序列与HCV 1b基因亚型序列位于同一个进化分支,核苷酸同源性为96.8%~97.6%;1例病例序列与HCV 3a基因亚型序列位于同一个进化分支,核苷酸同源性为95.2%;1例病例序列与HCV 6n基因亚型序列位于同一个进化分支,核苷酸同源性为97.9%;1例病例与HCV 6u基因亚型序列位于同一个进化分支,核苷酸同源性为98.4%。结论 昆明地区存在着HCV 1b、HCV 3a、HCV 3b、HCV 6n和HCV 6u等多个基因型/基因亚型HCV流行,以HCV 3b为主要流行的基因亚型。
Objective To investigate the distribution characteristics of HCV genotypes and subtypes in patients with HIV (human immunodeficiency virus, HIV)/HCV co-infection in Kunming based on the nucleocapsid protein gene sequence of HCV (hepatitis C virus).Methods Serum was collected from HIV/HCV co-infected patients with household registration in 14 county-level cities, districts and counties under the jurisdiction of Kunming, who admitted to Yunnan Provincial Infectious Disease Hospital from March to August 2019. The viral RNA was extracted from the serum, reverse transcribed to synthesize cDNA, and the HCV nucleocapsid protein gene-specific primers were used for nested PCR amplification. The positive amplification products were sequenced, bioinformatics software such as DNAstar and MEGAX were used for sequence analysis.Results A total of 64 samples from co-infected patients with clinical diagnosis of suspected HIV/HCV were collected and amplified by HCV nucleocapsid protein gene-specific primers, of which 17 samples were amplified positively. The results of sequence analysis showed that the sequences of 9 cases were located in the same evolutionary branch as the HCV 3b subtype sequence, and the nucleotide homology was 93.3%-95.2%; the sequences of 5 cases were located in the same evolutionary branch as the HCV 1b subtype sequence, and the nucleotide homology was 96.8%-97.6%; the sequence of one case and the subtype sequence of HCV 3a gene were located in the same evolutionary branch, and the nucleotide homology was 95.2%; the sequence of one case and HCV 6n gene subtype sequence were located in the same evolutionary branch, and the nucleotide homology was 97.9%; One case was located in the same evolutionary branch as the HCV 6u gene subtype sequence, and the nucleotide homology was 98.4%.Conclusions HCV 1b, HCV 3a, HCV 3b, HCV 6n and HCV 6u genotypes or subtypes of HCV are prevalent in Kunming, and HCV 3b is the most prevalent genotype.
丙型肝炎病毒(hepatitis C virus, HCV)感染不仅可以引起肝脏炎症和肝细胞坏死, 更严重的是引起肝硬化以及肝癌的发生, 给人们身体健康带来严重的危害, 成为一个对人类健康危害较大的公共卫生问题。HCV为单股正链RNA病毒, 其基因组核苷酸序列全长约为9 600 bp, 编码3个结构蛋白和6个非结构蛋白[1]。根据基因序列, 目前在全世界已经确定有8种HCV基因型及100多种基因亚型, 基因型或基因亚型均有明显的地域性分布[2, 3]。中国以HCV 1b基因亚型最为常见(56.8%), 其次是HCV 2a基因亚型与基因3型, 而其他基因亚型较为少见。云南省位于中国西南, 其南部边境与缅甸、老挝、越南三国直接接壤, 且与东南亚多国相邻, 随着国际贸易和人口流动的增强, 国外流行病毒基因型或基因亚型传入的风险也在增加。云南目前存在HCV多个基因型或基因亚型病毒流行, 传染来源较为复杂。而不同基因型或基因亚型的HCV引起疾病严重程度、治疗效果存在明显差异[4]。因此本研究基于HCV 核衣壳蛋白基因对在云南省传染病医院入院的HIV共感染HCV患者中的HCV进行基因型或基因亚型分型研究, 了解昆明地区HIV/HCV共感染患者中的HCV基因型或基因亚型, 为临床上HIV/HCV共感染患者诊断、疾病严重程度的判断、抗病毒治疗提供依据。
2019年3— 8月在云南省传染病医院入院治疗的户籍地为昆明所辖14个县级市、区及县的HIV/HCV共感染患者, 所有HIV患者诊断均符合《中国艾滋病诊疗指南2019版》[5], HCV患者诊断符合《丙型肝炎防治指南(2019 年版)》标准[6], 同时收集患者个人信息。本研究通过昆明医科大学附属传染病医院伦理委员会审查。
1.2.1 血清样本采集 所有研究对象采用一次性采血针采集空腹8~12 h非抗凝血液5 mL, 静置30 min后, 3 000 r/min (离心半径为 8 cm)离心10 min, 取管中血样上层全部血清于1.5 mL离心管中, 血清样本采集后均保存于-80 ℃冰箱。
1.2.2 病毒RNA提取、cDNA合成及PCR扩增 采用病毒RNA提取试剂盒[TIANGAN, 天根生化科技(北京)有限公司]参照说明书的方法从患者血清样本中提取病毒RNA, 采用Reverse Transcriptase M-MLV(RNase H-)试剂[TAKARA, 宝生物工程(大连)有限公司]按照试剂盒说明书合成第一链cDNA。采用HCV核衣壳蛋白基因特异性引物HCF1/HCR1进行第一轮PCR扩增, 以2 μ L第一链合成cDNA为模板, 依次加入PCR Buffer(10 mmol/L)2 μ L、dNTP (2.5 mmol/L) 2 μ L、 ExTaq [TAKARA, 宝生物工程(大连)有限公司]0.3 μ L、上下游引物HCF1/HCR1各0.5 μ L, 12.7 μ L ddH2O; 采用HCF2/HCR2[7]为引物进行第2轮PCR扩增, 以2 μ L第1轮扩增产物为模板, 其他扩增体系与第1轮相似, 2轮扩增条件均相同为94 ℃ 5 min预变性; 94 ℃ 30 s, 55 ℃ 30 s , 72 ℃ 1 min, 35个循环; 最后72 ℃ 10 min。扩增产物用1% TAE琼脂凝胶电泳进行检测, 阳性扩增产物送生工生物工程(上海)股份有限公司昆明公司测序。
1.2.3 序列分析 采用DNAstar中SeqMan软件包进行序列拼接。在NCBI GeneBank中选取并下载不同国家地区的HCV 7个基因型30个基因亚型核衣壳蛋白基因序列, 同时下载昆明地区HCV核衣壳蛋白基因序列, 采用MAGA X软件中的Clustal W进行比对, 采用最大似然法(maximum likelihood)构建系统进化树, 筛选最佳DNA模型, Bootstrap值为1 000, 核苷酸同源性采用DNAstar中MegAlign进行分析。
共收集到64份临床诊断为HIV共感染HCV患者血清样本, 从血清样本中提取HCV-RNA, 采用HCV核衣壳蛋白基因特异性引物进行扩增检测, 其中17份样本阳性。这17份阳性病例男女比例为8∶ 9, 年龄(44.36± 5.85)岁, 患者均为汉族。
对PCR扩增阳性产物测序拼接后, 获得17条长364 nt序列。核苷酸同源性分析结果显示本研究的KM-0041、KM-0059、KM-0049、KM-0050、KM-0070 5例病例序列与所选取的HCV 1b基因亚型参考序列(EU256083)核苷酸序列同源性最高, 同源性为96.8%~97.6%, 而与所选取的其他基因亚型的参考序列核苷酸同源性均在85.6%以下, 提示这5例病例为HCV 1b基因亚型; 该5例病例核苷酸序列与20条2001— 2005年昆明地区流行的HCV 1b基因亚型序列的同源性为91.9%(DQ777809与KM-0070)~99.4%(EU081379、EU081383与KM-0050, EU081379、EU081383与KM-0059), 部分序列与本研究序列高度同源。KM-0090病例序列与所选取的HCV 3a基因亚型参考序列(KF035125)核苷酸序列同源性最高, 核苷酸同源性为95.2%, 而与所选取其他基因亚型参考序列的核苷酸同源性均在84.5%%以下, 提示该病例为HCV 3a基因亚型; 该病例核苷酸序列与2002— 2005年昆明地区2条HCV 3a序列的同源性为96.2%(DQ777805与KM-0090)~97.7%(EU081305与KM-0090)。KM-0043、KM-0051、KM-0054、KM-0058、KM-0063、KM-0065、KM-0072、KM-0080和KM-0088共9例病例序列与所选取的HCV 3b基因亚型参考序列(LC368420)核苷酸序列较高, 核苷酸同源性为93.3%(KM-0063)~95.2%(KM-0051、KM-0072、KM-0080), 与所选取的其他基因亚型参考序列的核苷酸同源性均在81.6%以下; 该9例病例核苷酸序列与2001— 2005年昆明地区16条HCV 3b基因亚型的核苷酸同源性为92.2%(KM-0058与DQ777785)~99.1%(KM-0065与DQ777783、KM-0072与DQ777800、KM-0065与EU081384)。KM-0066病例序列与所选取的HCV 6u基因亚型参考序列(EU-408332)核苷酸同源性最高, 为98.4%, 而与所选取其他基因亚型参考序列的同源性均在86.9%以下, 提示该病例为HCV 6u基因亚型; KM-0061病例核苷酸序列与所选取的HCV 6n基因亚型参考序列(AY878652)的核苷酸同源性最高, 为97.9%, 与所选取的其他基因亚型参考序列的核苷酸同源性均在91.2%以下, 提示该病例为HCV 6n基因亚型; 该病例核苷酸序列与2001— 2005年昆明地区5条HCV 6n基因亚型核苷酸序列同源性为90.5%(DQ777780与KM-0061)~98.8%(EU081310与KM-0061)。见表1。
| 表1 本研究毒株序列与参考序列的核苷酸同源性 % Table 1 Nucleotide homology between the reference sequence and sequence of this study % |
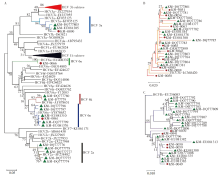
将本研究中获得17条序列与NCBI GeneBank中下载的50条昆明地区和30条其他地区不同基因亚型的HCV核衣壳蛋白基因核苷酸序列一起构建遗传进化树。遗传进化分析结果显示(图1), KM-0041、KM-0049、KM-0050、KM-0059、KM-0070等5条序列与20条2001— 2005年昆明地区流行的以及选取其他地区的1b型HCV位于同一个进化分支内, 进一步分析发现所有1b型HCV形成3个较小的进化簇, 本次研究的5条与2001— 2004年昆明地区HIV/HCV共感染6条序列集中在第1进化簇中; KM-0090序列与2条2001— 2005年昆明地区流行的和选取其他地区的3a型HCV位于同一个进化分支; KM-0043、KM-0051、KM-0054、KM-0058、KM-0063、KM-0065、KM-0072、KM-0080、KM-0088 9条序列与16条2001— 2005年昆明地区流行的和选取其他地区3b型HCV位于同一个进化分支; KM-0061序列与5条2001— 2005年昆明地区流行的和选取其他地区6n型HCV位于同一个进化分支; KM-0066序列与所选取的HCV 6u参考株(EU408332)位于同一个进化分支。
云南省是我国艾滋病和丙型肝炎流行最严重的地区, 而这两种病毒共感染率非常高。昆明市是云南省的省会, 人口密度高, 人员流动大, HIV/HCV共感染率也很高。目前在昆明地区HIV/HCV共感染人群中已经存在1b、2a、3a、3b、6a、6b、 6n等基因亚型HCV流行, 以3b 、1b基因亚型HCV流行为主[8, 9, 10]。昆明地区HCV基因型或基因亚型的流行分布也呈现多样性的特点[11, 12]。昆明市HCV感染人群中不仅存在本研究中检测到的HCV 1b、HCV 3a、HCV 3b、HCV 6n 4个基因亚型病毒[13, 14, 15], 既往的研究还发现2a和6b基因亚型病毒[8, 15], 而且还发现存在6v、6k两个稀有的基因亚型 [14, 16, 17]。2008年的研究表明[16], 2008年以前昆明地区HCV患者主要以HCV 1b基因亚型为主, 其次为HCV基因3型, 而2010— 2016的研究表明[9, 10, 14], HCV基因亚型以HCV 3b基因为主, 其次为HCV 3a基因亚型, HCV 1b基因亚型的比重相比较2008年前有所下降。本研究结果表明, 2019年昆明地区HCV主要以HCV 3b基因亚型为主, 与2010— 2016年间的分布相符, 提示昆明地区HCV患者基因型分布自2010年以来始终保持相对稳定, 昆明地区的主要流行基因型或基因亚型未发生大的变化。
虽然本次研究在昆明地区HIV共感染人群中未发现2a、6a、6b基因亚型HCV, 但在该地区HIV/HCV共感染人群中新发现且仅在边境地区流行[11]的6u基因亚型病毒, 提示在昆明地区HIV/HCV共感染人群存在多个基因亚型HCV流行, 而且某些基因亚型HCV的流行分布可能发生了改变。昆明地区HCV出现这种多基因亚型共同流行, 而以3型和6型HCV流行为主的流行方式的原因是:由于昆明地区处于我国内地以1型和2型HCV流行为主的地区[18]与东南亚以3型和6型为主的地区交汇处, 因此存在这两个地区基因型病毒共同流行的态势。
云南省位于我国西南, 与东南亚国家直接接壤或相邻, 东南亚国家流行的HCV 基因型主要以6型为主[19], 随着近年来国际贸易和交流的增多, 导致我省边境地区不断发现基因6型多个基因亚型病毒流行, 甚至发现基因6型新的基因亚型病毒6u、6k病毒流行[20, 21]。本次研究在昆明地区不仅发现存在6n基因亚型HCV流行, 更为重要的是在昆明地区首次发现了存在6u基因亚型HCV流行, 提示基因6型HCV流行范围有不断扩大的趋势, 因此今后在昆明地区需要加强基因6型HCV监测和检测。
与云南省HCV的流行相似, 昆明地区HCV基因型或基因亚型的流行分布也呈现多样性的特点[1, 2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12]。本研究在昆明地区HIV/HCV共感染人群中发HCV 1b、HCV 3a、HCV 3b、HCV 6n 4个单纯性HCV感染人群基因亚型病毒[13, 14, 15], 同时也检测到罕见的HCV6u基因亚型。既往研究发现, 在昆明地区HIV/HCV共感染人群中还存在HCV 2a、6b、6v、6k等基因亚型病毒[8, 14, 15, 16, 17]。此外, 本研究发现昆明地区HIV/HCV人群中主要流行病毒基因亚型与该地区单纯性HCV感染人群中主要流行病毒基因亚型相似[14], 提示昆明地区HCV单感染人群与HIV/HCV共感染人群中HCV患者基因亚型分布均呈现多基因型与多基因亚型分布的特点。研究表明[22], 云南地区单纯感染HCV人群及HIV/HCV共感染人群中HCV基因亚型均以HCV 3b基因亚型为主, 其次为HCV 3a基因亚型, 单纯感染HCV人群与HIV/HCV共感染人群主要流行基因亚型不存在差异。昆明地区HCV基因亚型分布, 也存在同样的情况, 昆明地区HCV单感染患者中主要以HCV3b基因亚型为主, HCV 3a基因亚型也占很大比例[10, 17], 而昆明地区HIV/HCV共感染人群HCV基因亚型主要以HCV3b基因亚型为主[9], 与本研究的结果一致, 提示云南省地区, 昆明地区HCV单感染人群与HIV/HCV共感染人群中HCV流行的主要基因亚型为HCV 3b与HCV 3a基因亚型, 其中主要以HCV 3b基因亚型为主。
云南省HIV感染人群中共感染HCV非常严重[23], 两种病毒共感染后, 加重HCV相关肝病严重程度[24, 25], 导致HIV感染者死亡率增加, 成为云南省一个严重的公共卫生问题。本次研究结果提示昆明地区HIV共感染人群中存在多个基因亚型HCV流行, 传染来源非常复杂, 给临床上HIV共感染HCV患者的诊断、疾病严重程度的判断、抗病毒治疗带来很大困难。由于本次研究仅对昆明地区1家医院进行了6个月入院病例的研究, 所研究样本数量和监测的时间相对较短, 尚不能完全代表整个昆明地区基因分型情况, 特别是近年来多条通往东南亚国家的铁路和高速公路的通车, 人员流动增多, HCV传播扩散速度可能加快, 因此需要进一步加强昆明及周边地区多家医院HIV共感染患者中HCV基因型或基因亚型的监测, 这对昆明地区乃至云南省临床上HIV共感染HCV患者诊断、抗病毒治疗及鉴定传染源、追溯传染途径具有重要意义。
利益冲突声明 所有作者声明不存在利益冲突
编辑:王佳燕
| [1] |
|
| [2] |
|
| [3] |
|
| [4] |
|
| [5] |
|
| [6] |
|
| [7] |
|
| [8] |
|
| [9] |
|
| [10] |
|
| [11] |
|
| [12] |
|
| [13] |
|
| [14] |
|
| [15] |
|
| [16] |
|
| [17] |
|
| [18] |
|
| [19] |
|
| [20] |
|
| [21] |
|
| [22] |
|
| [23] |
|
| [24] |
|
| [25] |
|